4.2.7 Коннектомика сегодня
Чего не могу воссоздать, того не понимаю.
Ричард Фейнман
EyeWire не единственный из современных проектов, посвящённых развитию технологий для создания моделей коннектомов живых существ (напоминаем, что коннектом — это карта связей нейронов в нервной ткани).
Второго апреля 2013 г. администрация Президента США дала старт амбициозной частно-государственной исследовательской программе BRAIN Initiative (Brain Research through Advancing Innovative Neurotechnologies, Исследование мозга через продвижение инновационных нейротехнологий), глобальной целью которой является достижение динамического понимания принципов работы мозга. Инициатива была разработана Управлением по научной и технологической политике (Office of Science and Technology Policy, OSTP) в рамках более широкой программы Neuroscience Initiative (Инициативы нейронауки)[1]. Созданная под впечатлением от проекта «Геном человека» (Human Genome Project) BRAIN Initiative среди своих публичных целей заявляет помощь исследователям в деле изучения мозговых расстройств, таких как болезни Альцгеймера и Паркинсона, депрессии и травматических поражений мозга.
Первым кирпичиком в фундаменте BRAIN Initiative стала статья Павлоса (Пола) Аливизатоса из Калифорнийского университета в Беркли и его коллег, вышедшая на страницах престижного журнала Neuron под названием «Проект карты активности мозга и проблема функциональной коннектомики» (The Brain Activity Map Project and the Challenge of Functional Connectomics) за год до старта проекта. В статье были изложены экспериментальные планы для более скромного проекта, в том числе рассмотрены методы, которые могут быть использованы для построения «функционального коннектома», а также перечислены технологии, которые необходимо будет разработать в ходе проекта. Авторы указывают, что первоначальные исследования могут быть проведены на излюбленных объектах исследователей — нематодах Caenorhabditis elegans, затем на плодовых мушках Drosophila melanogaster — обладателях сравнительно простых нервных систем. На следующем этапе исследований предлагалось перейти к рыбкам Danio rerio (известных в англоязычной литературе под именем Zebrafish — рыба-зебра), мышам и (ВНЕЗАПНО!) этрусской землеройке (она же карликовая многозубка)[2].
Кстати сказать, эта чудесная многозубка — самое маленькое по массе тела из известных науке млекопитающих (если брать в качестве критерия длину тела, то свиноносая летучая мышь оказывается всё же немного покороче), а её мозг состоит из всего примерно миллиона нейронов. Сердечко этрусской землеройки бьётся с частотой до 1511 ударов в минуту[3]. А с какой частотой билось бы твоё сердце, %USERNAME%, если бы ты узнал, что учёные хотят нарезать твой мозг на тонкие ломтики?
От землероек можно будет перейти к приматам, в том числе — на последнем этапе — к людям. Среди технологий, предлагаемых для разработки авторами статьи: наночастицы, которые могли бы использоваться в качестве датчиков, способных определять потенциалы действия в отдельных нейронах; нанозонды, которые могли бы служить в качестве электрофизиологических многоэлектродных массивов; многообещающие методы, основанные на синтетической биологии, и множество других прекрасных гитик[4].
Интересен мультидисциплинарный состав авторов этого своеобразного манифеста. Аливизатос — химик и пионер в области развития наноматериалов, Миянг Чунь — биохимик, Джордж Чёрч — генетик, химик и молекулярный инженер, Ральф Гринспен и Рафаэль Юст — нейробиологи, Майкл Рукс — физик.
Что сделано сегодня из обозначенного в статье 2012-го? Не так уж мало.
Первый коннектом живого существа, а именно нематоды C. elegans, был построен в далёком 1986 г. группой исследователей во главе с биологом Сидни Бреннером из Кембриджа. Бреннер и его коллеги аккуратно нарезали миллиметровых червей на тонкие ломтики и сфотографировали каждый срез с помощью плёночной камеры, установленной на электронном микроскопе, а затем по полученным снимкам вручную проследили все связи между нейронами[5]. Однако у C. elegans всего 302 нейрона и около 7600 синапсов. В 2016 г. команда учёных из Университета Дэлхаузи в Канаде повторила подвиг своих коллег для личинки морского оболочника Ciona intestinalis, центральная нервная система которого, как выяснилось, состояла из 177 нейронов и 6618 синаптических соединений[6]. Однако надо заметить, что методы, используемые для построения коннектома, неэффективны для крупных нервных систем. Исследователи не задумывались всерьёз о том, чтобы приступить к осуществлению значительно более крупных проектов до 2004 г., когда физик Винфрид Денк и нейроанатом Хайнц Хорстманн из Института медицинских исследований Общества Макса Планка предложили новый метод, основанный на использовании автоматического микроскопа для разрезания и визуализации мозга, а также специализированного программного обеспечения для сбора и соединения результирующих изображений[7].
В 2019 г. в журнале Nature появилась публикация доктора Скотта Эммонса и его коллег из Медицинского колледжа Альберта Эйнштейна (Albert Einstein College of Medicine) с подробным отчётом о воссоздании коннектома обоих полов (гермафродита и самца) нематоды C. elegans при помощи вышеуказанного метода[8]. Годом раньше группа учёных под руководством Чжихао Чжэна из Принстонского университета завершила работу над сканированием мозга дрозофилы, состоящего из примерно 100 000 нейронов. Система, разработанная Чжэном и его коллегами, позволила пропустить через просвечивающий растровый электронный микроскоп более 7000 тончайших срезов мозга мушки, толщина каждого из которых составляла порядка 40 нм, а суммарный размер полученных в результате изображений составил 40 трлн пикселей[9], [10].
Пятого августа 2019 г. команда, в которую, помимо Чжэна, входили специалисты из исследовательской группы Connectomics компании Google, а также их коллеги из Медицинского института Говарда Хьюза (HHMI) и Кембриджского университета, опубликовала основанную на собранных годом ранее данных работу «Автоматическая реконструкция мозга дрозофилы на базе электронной микроскопии последовательных сечений с помощью закрашивающих сетей и локальной коррекции выравнивания» (Automated Reconstruction of a Serial-Section EM Drosophila Brain with Flood-Filling Networks and Local Realignment), в которой демонстрируется метод, способный с использованием нейросетевой модели автоматически воссоздавать объёмную карту связей нейронов[11]. Запись в блоге Google AI, опубликованная за год до выхода статьи, посвящённой реконструкции мозга дрозофилы, показывает работу алгоритма на примере мозга зебровой амадины (Taeniopygia guttata)[12], которая по-английски называется Zebra finch. Быть может, какой-то стажёр из Google перепутал Zebrafish с Zebra finch, а в эти минуты уже подкрадывается с микротомом к кубинскому щелезубу, перепутав его с карликовой многозубкой?
Рис. 87. Карликовая многозубка (лат. Suncus etruscus)
В апреле 2019 г. сотрудники Алленовского института головного мозга в Сиэтле отпраздновали преодоление последнего рубежа в проекте по картированию одного кубического миллиметра мозга мыши с его 100 000 нейронов и одним миллиардом связей между ними. Чтобы обработать образец размером с горчичное зёрнышко, микроскопы работали непрерывно в течение пяти месяцев, собрав более 100 млн изображений 25 000 срезов зрительной коры. Затем программному обеспечению, разработанному учёными института, потребовалось около трёх месяцев, чтобы объединить изображения в единый трёхмерный массив объёмом 2 петабайта (т. е. 2 млн гигабайт). Все собранные более чем за тридцать лет миссиями Landsat снимки нашей планеты занимают всего около 1,3 петабайта, что делает сканы мозга мыши практически «целым миром в песчинке», говорит нейробиолог Клэй Рэйд, обыгрывая слова английского поэта Уильяма Блейка [To see a World in a Grain of Sand].
Первого июня 2021 г. в исследовательском блоге компании Google появилось сообщение[13], что её исследователи совместно с коллегами из Лаборатории Лихтмана (Lichtman Laboratory) в Гарвардском университете опубликовали датасет под названием H01. Этот набор данных объёмом 1,4 петабайта содержит информацию о небольшом фрагменте коры головного мозга человека. Ряд технических подробностей можно узнать из сопроводительной статьи под названием «Коннектомное исследование петамасштабного фрагмента коры мозга человека» (A connectomic study of a petascale fragment of human cerebral cortex)[14], вышедшей днём ранее на сайте препринтов bioRxiv.
В ходе хирургической операции из височной доли коры головного мозга 45-летней пациентки, страдающей от устойчивой к медикаментам эпилепсии, был извлечён положенный в основу исследования фрагмент ткани объёмом около кубического миллиметра. При помощи микротома этот фрагмент был разделён более чем на 5000 срезов толщиной 30 нм, а затем обработан многолучевым сканирующим электронным микроскопом с разрешением 4 × 4 нм. После этого полученные данные были превращены в трёхмерную модель при помощи уже ранее знакомых нам закрашивающих сетей (Flood-Filling Networks, FFNs) и вспомогательных вычислительных моделей. Полученный датасет содержит детальные сведения о 50 000 клеток, сотнях миллионов их отростков (авторы статьи употребляют термин «нейриты» — собирательное понятие для аксонов и дендритов) и примерно 130 млн синапсов. На сегодняшний день H01 является крупнейшим размеченным набором данных, описывающим образец мозговой ткани человека.
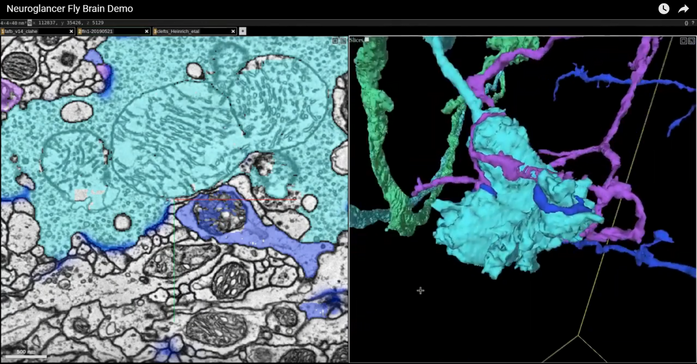
Ознакомиться с данными можно в обычном браузере при помощи специального интерфейса, получившего название Neuroglancer (дословно: нейронаблюдатель)[15].
Рис. 88. Трёхмерная модель мозга в Neuroglancer
Ещё одним важным результатом, полученным в последнее десятилетие, стало создание трёхмерной модели синапса в атомарном разрешении, включающей около 300 000 молекул, принадлежащих к 60 различным белкам[16], [17].
Хотя в ходе упомянутых исследований и достигнут несомненный прогресс, конечная цель — наноразмерный коннектом человеческого мозга — пока ещё далеко. Число нейронов в нём сопоставимо с количеством звёзд в Млечном Пути (порядка 1011). При использовании современной технологии обработки изображений потребуются десятки микроскопов, работающих круглосуточно на протяжении тысячи лет, чтобы собрать данные, необходимые для достижения конечной цели.
Удивительно, что лишь недавно удалось обнаружить некоторые новые типы клеток мозга, а также уточнить функции известных ранее клеток. Например, в 2015 г. учёные из Федеральной политехнической школы Лозанны (École Polytechnique Fédérale de Lausanne) разработали количественную модель ранее неизвестной взаимосвязи между астроцитами[18] и нейронами[19]. А в 2023 г. учёным из Лозаннского университета (Université de Lausanne) удалось обнаружить новую разновидность специализированных астроцитов, участвующих в переносе одного из нейромедиаторов — глутаминовой кислоты (глутамата)[20]. Словом, в 2020-е гг. рубрика «Новости анатомии» всё ещё не является курьёзным анахронизмом.
Но достижения в области микроскопии, а также разработка более мощных компьютеров и алгоритмов для анализа изображений продвинули область коннектомики вперёд столь быстро, что это удивляет и самих исследователей. «Пять лет назад было слишком амбициозно думать о кубическом миллиметре», — говорил Рэйд в 2019 г. Сегодня многие исследователи считают, что полное картирование мозга мыши, объём которого составляет около 500 кубических миллиметров, станет возможным уже в этом десятилетии. «Сегодня картирование человеческого мозга на синаптическом уровне может показаться невероятным. Но если прогресс в вычислительных мощностях и в научных методах будет идти вперёд теми же темпами, ещё одно тысячекратное увеличение возможностей уже не кажется нам немыслимым»[21].
BRAIN Initiative — не единственная масштабная программа в этой области. Созданием функциональной модели мозга крысы (с прицелом на мозг человека) заняты и учёные из проектов Blue Brain Project и Human Brain Project. Не стоит на месте и China Brain Project. Пожалуй, при взгляде со стороны сегодняшние усилия в области моделирования мозга напоминают космическую гонку середины XX в.
- ↑ * Нейронаука — междисциплинарная область знаний, занимающаяся изучением нейронных процессов.
- ↑ Alivisatos P. A., Chun M., Church G. M., Greenspan R. J., Roukes M. L., Yuste R. (2012). The Brain Activity Map Project and the Challenge of Functional Connectomics / Neuron, Vol. 74, Iss. 6, pp. 970—974, June 21, 2012 // https://doi.org/10.1016/j.neuron.2012.06.006
- ↑ Jürgens K. D. (2002). Etruscan shrew muscle: the consequences of being small. The Journal of Experimental Biology. 205 (Pt 15): 2161–2166 // https://www.ncbi.nlm.nih.gov/pubmed/12110649
- ↑ Alivisatos P. A., Chun M., Church G. M., Greenspan R. J., Roukes M. L., Yuste R. (2012). The Brain Activity Map Project and the Challenge of Functional Connectomics / Neuron, Vol. 74, Iss. 6, pp. 970—974, June 21, 2012 // https://doi.org/10.1016/j.neuron.2012.06.006
- ↑ White J. G., Southgate E., Thomson J. N., Brenner S. (1986). The structure of the nervous system of the nematode Caenorhabditis elegans / Philosophical Transactions of the Royal Society B, Vol. 314, Iss. 1165, 12 November 1986, pp. 1—340 // https://doi.org/10.1098/rstb.1986.0056
- ↑ Ryan R., Lu Z., Meinertzhagen I. A. (2016). The CNS connectome of a tadpole larva of Ciona intestinalis (L.) highlights sidedness in the brain of a chordate sibling / eLife 2016; 5:e16962 // https://doi.org/10.7554/eLife.16962
- ↑ DeWeerdt S. (2019). How to map the brain / Nature, Vol. 571, S6-S8, 24 July 2019 // https://www.nature.com/articles/d41586-019-02208-0
- ↑ Cook S. J., Jarrell T. A., Brittin C. A., Wang Y., Bloniarz A. E., Yakovlev M. A., Nguyen K. C. Q., Tang L. T.-H., Bayer E. A., Duerr J. S., Bülow H. E., Hobert O., Hall D. H., Emmons S. W. (2019). Whole-animal connectomes of both Caenorhabditis elegans sexes / Nature, Vol. 571, pp. 63—71 // https://doi.org/10.1038/s41586-019-1352-7
- ↑ Zheng Z., Lauritzen J. S., Perlman E., Robinson C. G., Nichols M., Milkie D., Torrens O., Price J., Fisher C. B., Sharifi N., Calle-Schuler S. A., Kmecova L., Ali I. J., Karsh B., Trautman E. T., Bogovic J. A., Hanslovsky P., Jefferis G. S. X. E., Kazhdan M., Khairy K., Saalfeld S., Fetter R. D., Bock D. D. A Complete Electron Microscopy Volume of the Brain of Adult Drosophila melanogaster / Cell, Vol. 174, Iss. 3, pp. 730—743.E22, July 26, 2018 // https://doi.org/10.1016/j.cell.2018.06.019
- ↑ Li P. H., Maitin-Shepard J. (2019). An Interactive, Automated 3D Reconstruction of a Fly Brain / Google AI Blog, August 5, 2019 // https://ai.googleblog.com/2019/08/an-interactive-automated-3d.html
- ↑ Li P. H., Lindsey L. F., Januszewski M., Zheng Z., Bates A. S., Taisz I., Tyka M., Nichols M., Li F., Perlman E., Maitin-Shepard J., Blakely T., Leavitt L., Jefferis G. S. X. E., Bock D., Jain V. (2019). Automated Reconstruction of a Serial-Section EM Drosophila Brain with Flood-Filling Networks and Local Realignment // https://doi.org/10.1101/605634
- ↑ Jain V., Januszewski M. (2018). Improving Connectomics by an Order of Magnitude / Google AI Blog, July 16, 2018 // https://ai.googleblog.com/2018/07/improving-connectomics-by-order-of.html
- ↑ Blakely T. (2021). A Browsable Petascale Reconstruction of the Human Cortex / Google AI Blog, June 1, 2021 // https://ai.googleblog.com/2021/06/a-browsable-petascale-reconstruction-of.html
- ↑ Shapson-Coe A., Januszewski M., Berger D. R., Pope A., Wu Y., Blakely T., Schalek R. L., Li P., Wang S., Maitin-Shepard J., Karlupia N., Dorkenwald S., Sjostedt E., Leavitt L., Lee D., Bailey L., Fitzmaurice A., Kar R., Field B., Wu H., Wagner-Carena J., Aley D., Lau J., Lin Z., Wei D., Pfister H., Peleg A., Jain V., Lichtman J. W. (2021). A connectomic study of a petascale fragment of human cerebral cortex // https://doi.org/10.1101/2021.05.29.446289
- ↑ Explore H01: One cubic millimeter of the human cerebral cortex (2021) // https://h01-release.storage.googleapis.com/explore.html
- ↑ Wilhelm B. G., Mandad S., Truckenbrodt S., Kröhnert K., Schäfer C., Rammner B., Koo S. J., Claßen G. A., Krauss M., Haucke V., Urlaub H., Rizzoli S. O. (2014). Composition of isolated synaptic boutons reveals the amounts of vesicle trafficking proteins. / Science, Vol. 344, Iss. 6187, pp. 1023—1028 // https://doi.org/10.1126/science.1252884
- ↑ Doerr A. (2014). Modeling the synapse / Nature Methods, Vol. 11, pp. 788–789 // https://doi.org/10.1038/nmeth.3057
- ↑ * Астроцит (от греч. άστρον — звезда и κύτος — клетка) — тип нейроглиальной клетки звёздчатой формы с многочисленными отростками.
- ↑ Jolivet R., Coggan J. S., Allaman I., Magistretti P. J. (2015). Multi-timescale Modeling of Activity-Dependent Metabolic Coupling in the Neuron-Glia-Vasculature Ensemble / PLOS Computational Biology, February 26, 2015. // https://doi.org/10.1371/journal.pcbi.1004036
- ↑ de Ceglia R., Ledonne A., Litvin D. G., Lind B. L., Carriero G., Latagliata E. C., Bindocci E., Di Castro M. A., Savtchouk I., Vitali I., Ranjak A., Congiu M., Canonica T., Wisden W., Harris K., Mameli M., Mercuri N., Telley L., Volterra A. (2023). Specialized astrocytes mediate glutamatergic gliotransmission in the CNS. / Nature, Vol. 262, 06 September 2023. // https://doi.org/10.1038/s41586-023-06502-w
- ↑ DeWeerdt S. (2019). How to map the brain / Nature, Vol. 571, S6-S8, 24 July 2019 // https://www.nature.com/articles/d41586-019-02208-0